I. Построить выравнивание заданного белка и белка-прототипа
с разметкой трансмембранных сегментов.
1. Сравнить нумерацию остатков белка-прототипа в UniProt
и PDB
По ID: 3H90 (транспортный белок) белка-прототипа FIEF_SERP5
из PDBsum
было получено выравнивание последовательности по данному
ID и последовательности P69380 (FIEF_ECOLI) изUniProt, которая
затем была переведена в fasta формат. После чего выравнивание
было импортировано в GenDoc и сохранено: algn1.msf
Нумерация выравниваний последовательностей совпадает.
2. Постройте полное глобальное выравнивание заданного
белка и белка-прототипа.
Получив последовательности белков FIEF_ECOLI.fasta
и FIEF_SERP5.fasta было произведено
их выравнивание командой:
needle FIEF_ECOLI.fasta FIEF_SERP5.fasta align.needle -auto
Выходной файл: align.needle
Характеристики выравнивания:
# Length: 302
# Identity: 235/302 (77.8%)
# Similarity: 273/302 (90.4%)
# Gaps: 4/302 ( 1.3%)
# Score: 1231.5
3. Создать по данным БД ОРМ разметку
трансмембранных сегментов в белке-прототипе.
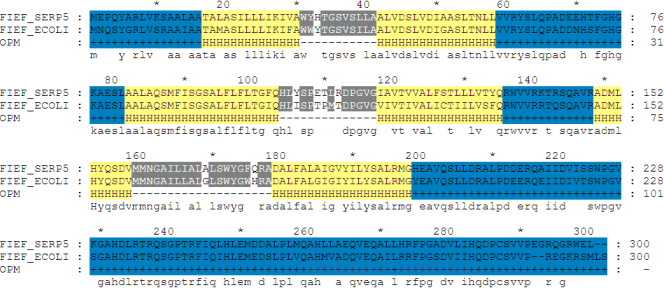
На синем фоне цитоплазматический участок
На желтом трансмембранный.
mark.msf
4. Предсказать топологию заданного белка с помощью наиболее
популярной программы (TMHMM).
Предсказанная топология заданного белка FIEF_SERP5 с помощью
сервера TMHMM - результат
mark2.msf файл с добавленной
последовательностью с разметкой трансмембранных спиралей
в соответствии с результатами предсказания:
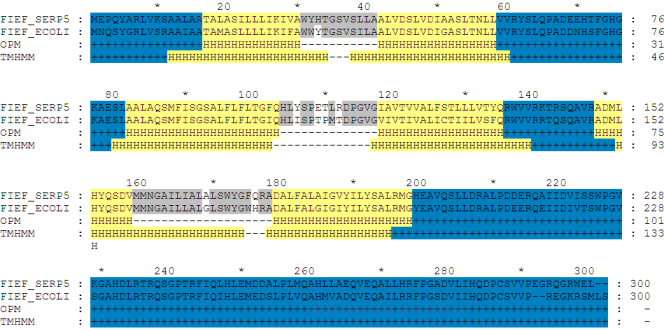
II. Сравнение полученного предсказания с данными ОРМ.
Результаты предсказания топологии мембранного белка FIEF_SERP5
| |
Число а.к. остатков
|
| Всего а.к. остатков |
300
|
| Остатки, предсказанные как локализованные
в мембране (всего) |
133
|
| Правильно предсказали (true positives,
TP) |
94
|
| Предсказали не то, что нужно (а.к. предсказаны
как мембранные, а по данным ОРМ таковыми не являются,
false positives, FP) |
39
|
| Правильно не предсказали ( не предсказаны,
и по данным ОРМ не находятся в мембране, true negatives,
TN) |
151
|
| Не предсказали то, что нужно (остатки
по данным ОРМ находятся в мембране, false negatives,
FN) |
7
|
| Чувствительность (sensivity) = TP / (TP+FN) |
0,93
|
| Специфичность (specificity) = TN
/ (TN+FP) |
0,79
|
| Точность(precision) = TP /(TP+FP) |
0,70
|
| Сверхпредсказание = FP/ (FP+TP) |
0,29
|
| Недопредсказание = FN / (TN+FN) |
0,044
|
Количесвто трансмембранных доменов, предсказанных TMHMM,
совпадает с количеством трансмембранных доменов ОРМ. И положение
приблизительно одинаково. Я считаю, что предсказание ТMHMM
довольно таки не плохое, о чем можно судить по проценту
недопредсказаных участков = 4,4%, однако процент сверхпредсказанных
участков = 29% говорит о том, что в данном случае программа
предпочла перезастраховаться и предсказала больше ТМ участков
чем было. В связи с этим точность находится на неидеальном
уровне = 70%.